清华新闻网6月17日电 在多细胞生物体中,尽管所有细胞共享相同的基因组,但由于内部基因调控网络的差异以及与周围微环境中相邻细胞的外部信号交流,它们在形态、基因表达和功能上展现出显著的多样性。相邻的不同种类细胞之间再通过复杂的相互作用,形成一定功能的组织模块。近年来,随着空间转录组技术的不断发展,研究者能够在单细胞分辨率下获得细胞的基因表达谱,同时保留细胞在组织内的空间位置信息。因此,如何有效地利用这些空间位置信息来揭示复杂组织中细胞的空间排列模式和相关的生物学功能,即识别空间细胞亚型和发现组织模块,是空间转录组数据分析的核心任务。
目前空间转录组数据分析面临以下两方面难题:第一,对于空间细胞类型的识别,许多研究仅使用细胞基因表达谱而忽视细胞的空间位置信息。然而,近年来的研究表明,原本被认为是同质性的细胞类型,根据其在组织中的位置,可以进一步细分为多个亚型。第二,对于组织模块的发现,当前的方法主要基于组织内区域的基因空间表达模式的一致性来发现组织模块。然而,构成组织的不同细胞的基因表达特征可能高度异质化,这些方法未能充分利用最新的单细胞分辨率的空间转录组数据中细胞类型的信息。
5月31日,太阳集团tyc5997生命科学学院/结构生物学高精尖创新中心/清华-北大生命科学联合中心张强锋副教授课题组在《细胞·系统》(Cell Systems)杂志在线发表题为“基于感知细胞-细胞相互作用的细胞嵌入在单细胞分辨率空间转录组数据中发现组织模块”(Tissue module discovery in single-cell resolution spatial transcriptomics data via cell-cell interaction-aware cell embedding)的研究论文。在该研究中,他们开发了基于图自编码器(Graph autoencoder)深度学习框架的人工智能算法SPACE,可以在空间转录组数据中识别空间细胞亚型和发现组织模块。
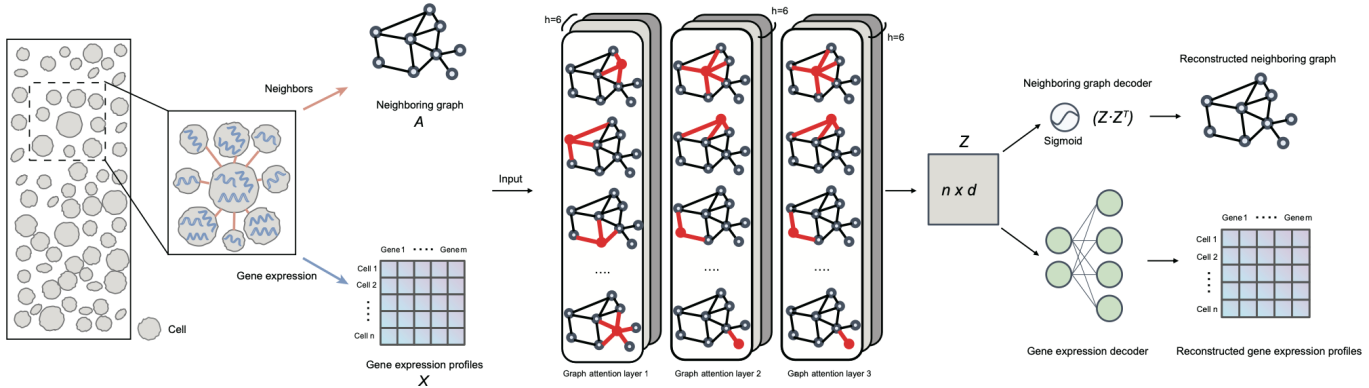
SPACE模型框架
SPACE使用图自编码器框架来学习低维的细胞嵌入,该细胞嵌入描述了空间转录组数据中每个细胞自身的基因表达信息以及其与空间邻近细胞的相互作用信息(因此称该细胞嵌入为细胞-细胞相互作用感知的细胞嵌入,cell-cell interaction-aware cell embedding)。在该细胞嵌入基础上,SPACE再通过聚类算法识别空间细胞亚型和发现组织模块。SPACE主要在以下两个方面区别于现有方法。首先,SPACE通过两个独立的解码器重构细胞自身的基因表达谱和空间邻近图。其次,SPACE定义了一个感知场比率α,该系数决定了基因表达谱重建损失与空间邻近图重构损失的相对权重。这种可调整的系数使SPACE能够将模型的优化重点(根据具体研究需要)转向强调所分析细胞自身的基因表达特征或空间邻近细胞的组织信息,从而识别空间细胞亚型或发现细胞群落,即一种具有可辨识边界且组成细胞类型空间分布的组织模块。通过使用多个空间转录组数据集对SPACE进行测试,可以表明SPACE发现的细胞群落与人工标注的组织结构在空间分布特征上相似。特别地,细胞群落是由其所组成的细胞间相似的近端相互作用网络而不是一致的基因空间表达模式来定义的。这种细胞间的近端相互作用网络可以用于优化基于配体-受体的细胞通讯推断,从而改进对生物过程中细胞间信号传递与调控的解读。
综上所述,研究人员开发了一个可以从单细胞分辨率的空间转录组数据中识别空间细胞类型和发现组织模块的空间转录组数据人工智能分析工具SPACE。SPACE可用于大规模的空间转录组研究,以了解空间邻近细胞之间的相互作用是如何影响细胞类型和组织模块生物学功能的。
张强锋为论文通讯作者,太阳集团tyc5997生命科学学院2019级博士生李雨哲和博士后张劲松为论文共同第一作者。沙特阿卜杜拉国王科技大学计算生物学科研中心主任,前百图生科(BioMap)研究院主任AI科学家高欣教授参与合作研究。
研究得到国家重点研究发展计划专项、国家自然科学基金、北京市结构生物学高精尖创新中心、清华-北大生命科学联合中心、太阳集团tyc5997计算平台、上海期智研究院和阿卜杜拉国王科技大学研究管理办公室的支持。
论文链接:
https://www.cell.com/cell-systems/fulltext/S2405-4712(24)00124-8
供稿:生命学院
编辑:李华山
审核:郭玲